Modelo de representación de superficies de estructuras macromoleculares
1
About :
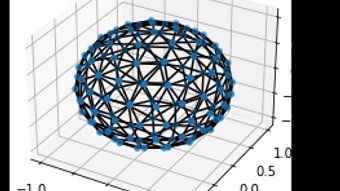
Algunas estructuras biológicas como macromoléculas, proteínas y plaquetas tienen cientos o miles de estructuras atómicas, donde las estructuras internas son irrelevantes a la hora de evaluar la interacción entre ellas. Al evaluar la interacción entre estructuras, la información presente en su superficie es suficiente, por esta razón, es importante contar con un modelo que abstraiga toda la estructura interna, manteniendo solo la presente en la superficie y evitando cálculos innecesarios.
Proponemos un módulo de Python nombrado pysurmolmesh para la construcción de un modelo representativo de las superficies de estructuras macromoleculares a través de una malla de partículas interactuantes que reflejan sus propiedades mecánicas y químicas, evitando contener información sobre las estructuras macromoleculares internas.
Tags :
Their limit for today is $0!



















Comments:
Reply:
To comment on this video please connect a HIVE account to your profile: Connect HIVE Account